
-

生物通官微
陪你抓住生命科技
跳動的脈搏
長讀長宏基因組組裝錯誤診斷與優化策略——四款主流工具評估
《Nature Biotechnology》:Troubleshooting common errors in assemblies of long-read metagenomes
【字體: 大 中 小 】 時間:2026年01月03日 來源:Nature Biotechnology 41.7
編輯推薦:
本研究針對長讀長宏基因組組裝準確性評估難題,開發了基于讀段剪切事件分析的開源工作流,系統評估了HiCanu、hifiasm-meta、metaFlye和metaMDBG四款工具在21個PacBio HiFi數據集上的表現。研究發現組裝錯誤率高達每1億堿基對46個錯誤,揭示多域嵌合體、過早環化等關鍵問題,為第三代測序技術在微生物基因組重建中的可靠性提供重要保障。
隨著第二代測序技術的發展,微生物基因組得以直接從環境宏基因組中重建,無需培養即可揭示微生物多樣性與功能。然而短讀長測序的局限性導致基因組組裝高度碎片化且存在污染。第三代測序技術(如PacBio和Oxford Nanopore)通過超長讀長突破重復序列限制,為復雜微生物群落研究帶來新機遇。但長讀長組裝算法在復雜環境中的應用準確性仍面臨挑戰,尤其對于缺乏參考基因組的低豐度微生物。
為系統評估長讀長宏基因組組裝質量,研究人員在《Nature Biotechnology》發表研究,選取HiCanu、hifiasm-meta、metaFlye和metaMDBG四款主流組裝工具,對包含模擬群落、腸道微生物組和海洋樣本的21個PacBio HiFi數據集進行基準測試。通過開發開源工具anvi-script-find-misassemblies量化讀段剪切事件(即長讀段在比對過程中被系統性分割的現象),首次實現組裝錯誤率的精確計量。
關鍵技術方法包括:使用minimap2進行長讀段與contig比對,通過anvi'o平臺構建contig數據庫并進行基因注釋,利用BLAST和k-mer分析驗證零覆蓋區域,結合pangenome(泛基因組)分析評估嵌合體錯誤。海洋樣本等新型生物群系數據的引入增強了結果的普適性。
組裝錯誤普遍存在于所有長讀長組裝工具
研究發現所有組裝工具均存在高置信度讀段剪切事件(100%剪切且覆蓋度≥10×)。metaMDBG在海洋樣本中產生的剪切事件比hifiasm-meta高三個數量級,錯誤率最高達每1億堿基對46個錯誤。零覆蓋區域(>1,000 bp)現象同樣普遍,metaMDBG中5.3%的contig存在此類問題。環化contig比例存在顯著工具間差異,metaMDBG報告的環化contig中最高77%存在剪切事件。
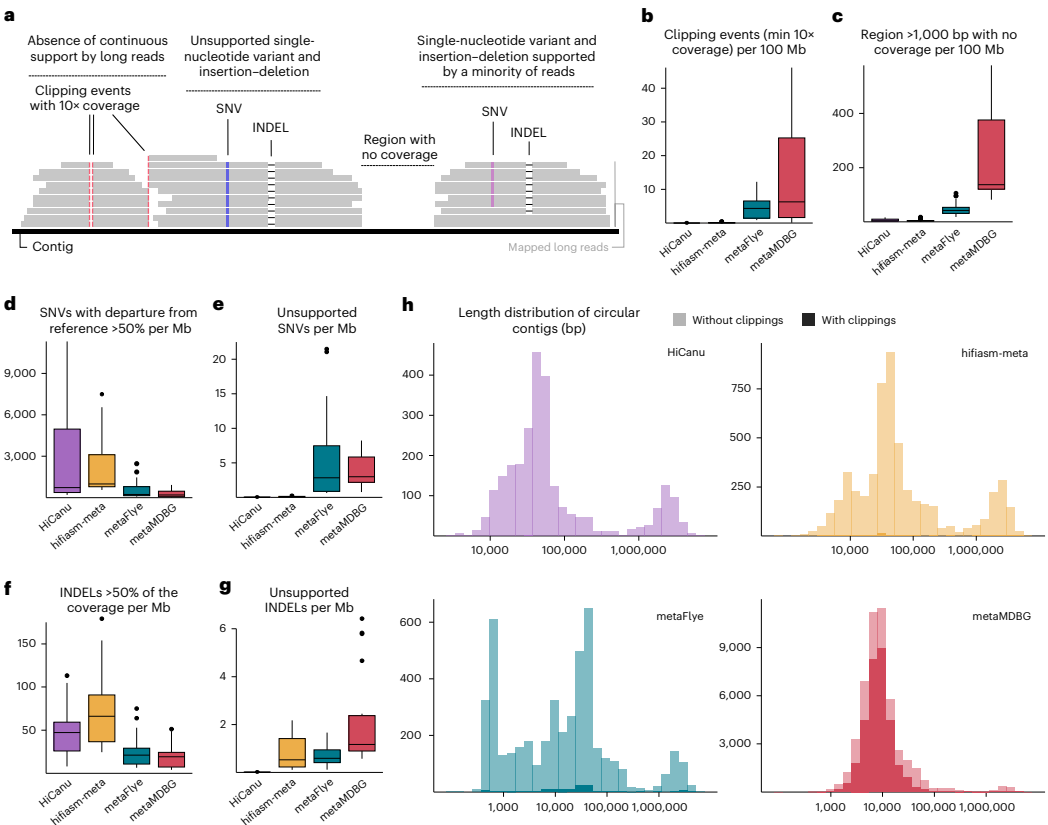
嵌合體contig
研究揭示多域嵌合體現象,如metaMDBG生成的contig同時包含廣古菌門、假單胞菌門、擬桿菌門和藍藻門序列。即便未觸發剪切警報,7.38 Mb的contig通過單拷貝核心基因(SCG)冗余度分析被發現拼接兩個拉氏菌科種群。雖然GC含量、覆蓋度突變等指標可輔助識別嵌合體,但大規模基因組研究中此類質量控制常被忽視。
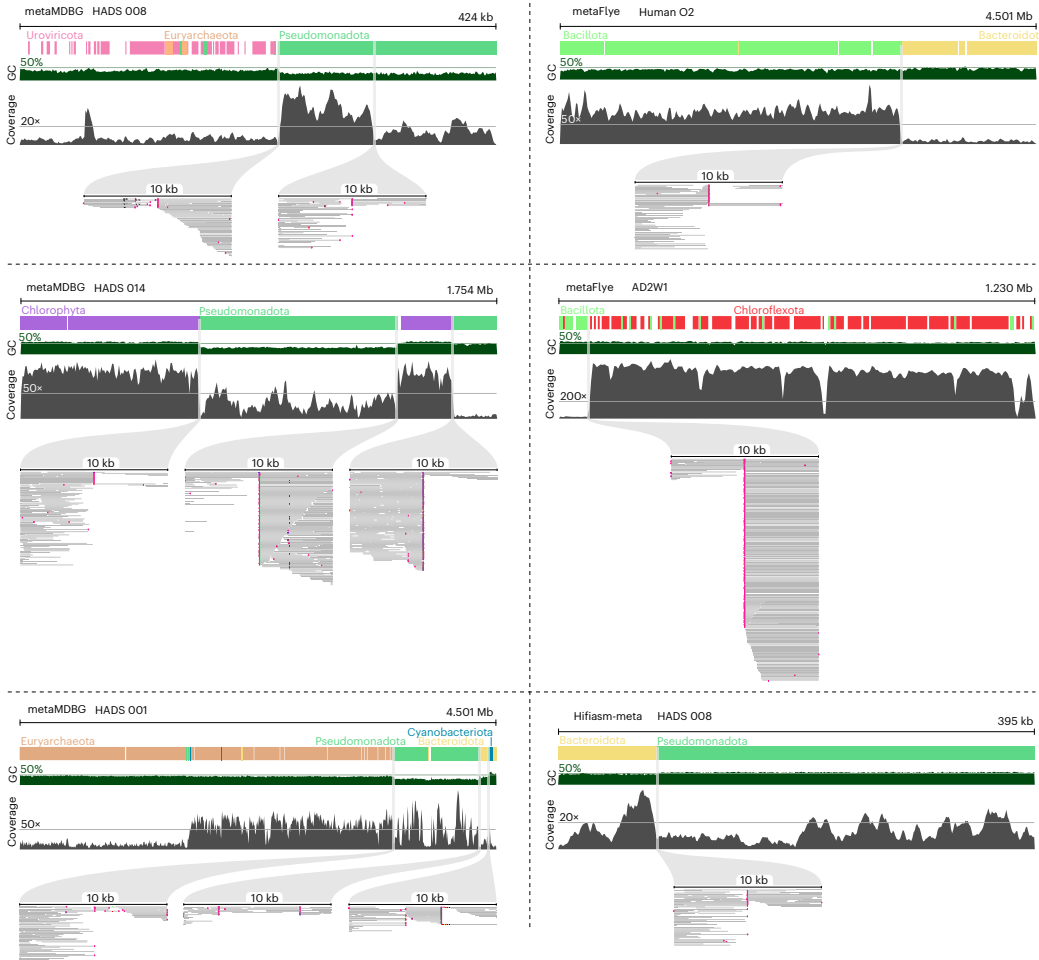
過早環化
環化contig報告機制存在嚴重可靠性問題。hifiasm-meta從厭氧消化池樣本中環化的甲烷絲菌屬基因組缺失甲烷生成關鍵代謝模塊,斷裂點位于轉座酶附近。通過設置保守篩選標準(環化contig<500 kb且含≥3個核糖體蛋白),發現metaMDBG的虛假環化率是hifiasm-meta的2倍、metaFlye的4倍。此類錯誤對質粒/病毒基因組重建產生嚴重影響。
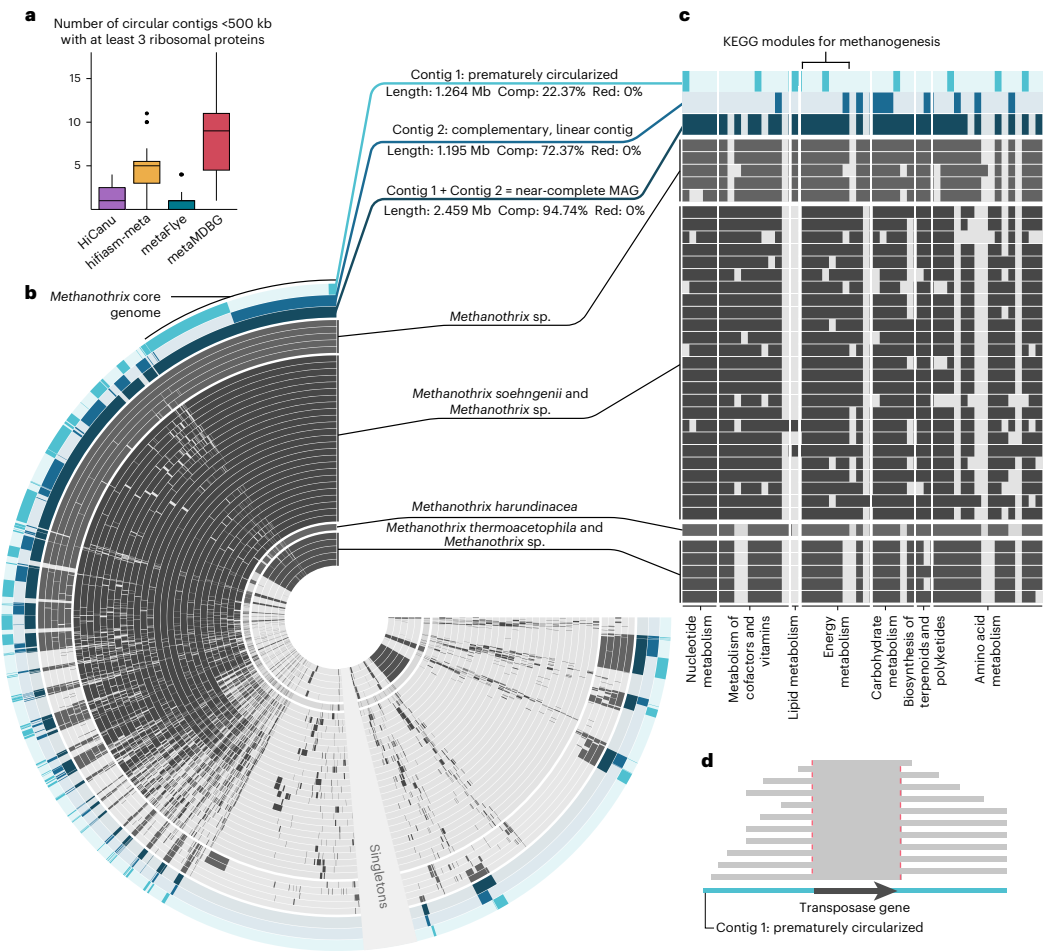
單倍型錯誤、虛假重復與幻影序列
研究報道三種單倍型解析錯誤:保守側翼區域與變異區錯誤拼接、次要單倍型序列被采納為主序列、無讀段支持的虛假重復。metaMDBG和metaFlye甚至產生超過5,000 bp的"幻影序列",其90%的k-mer(k=21)在原始讀段中缺失。這些錯誤會導致開放閱讀框(ORF)錯誤預測。
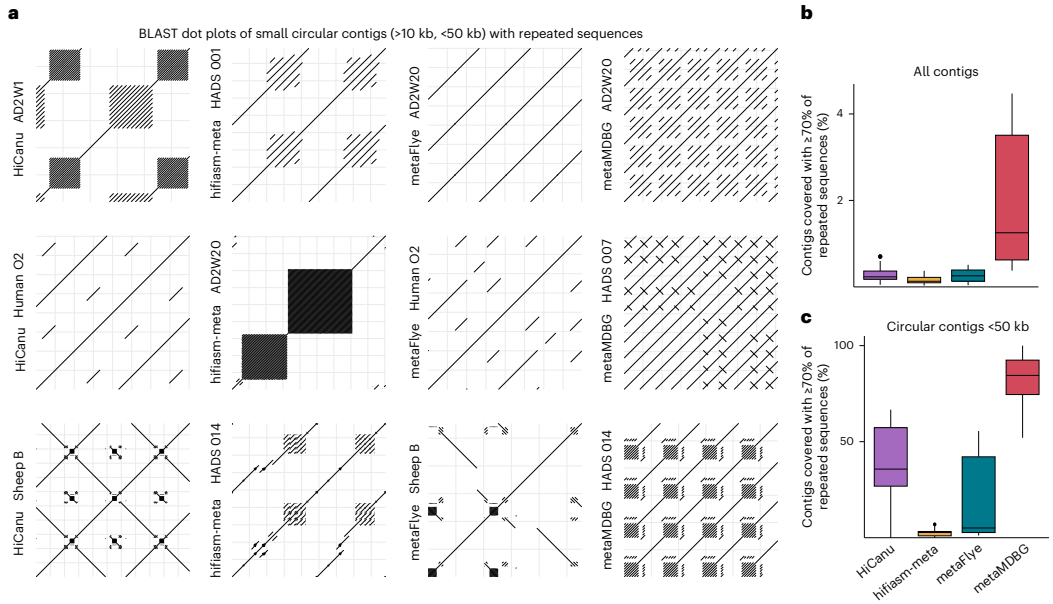
過度重復
自比對分析顯示,metaMDBG單個組裝可產生超過30萬個重復序列,最高重復長度達225,520 bp。海洋樣本中87%的環化contig(<50 kb)主要由重復序列構成,提示重復序列是虛假環化的誘因之一。天然重復與算法錯誤的區分需結合覆蓋度等多項指標。
模擬數據集的局限性
研究指出常用模擬數據集(如Zymo-HiFi D6331)無法反映真實樣本復雜性。hifiasm-meta在模擬數據中組裝規模異常膨脹(270 Mb vs 預期93 Mb),但在海洋樣本中表現優異,證明模擬數據預測力有限。對大腸桿菌菌株混合樣本的分析顯示,metaMDBG生成的"環化基因組"實為嵌合體,平均核苷酸一致性(ANI)比較會掩蓋此類錯誤。
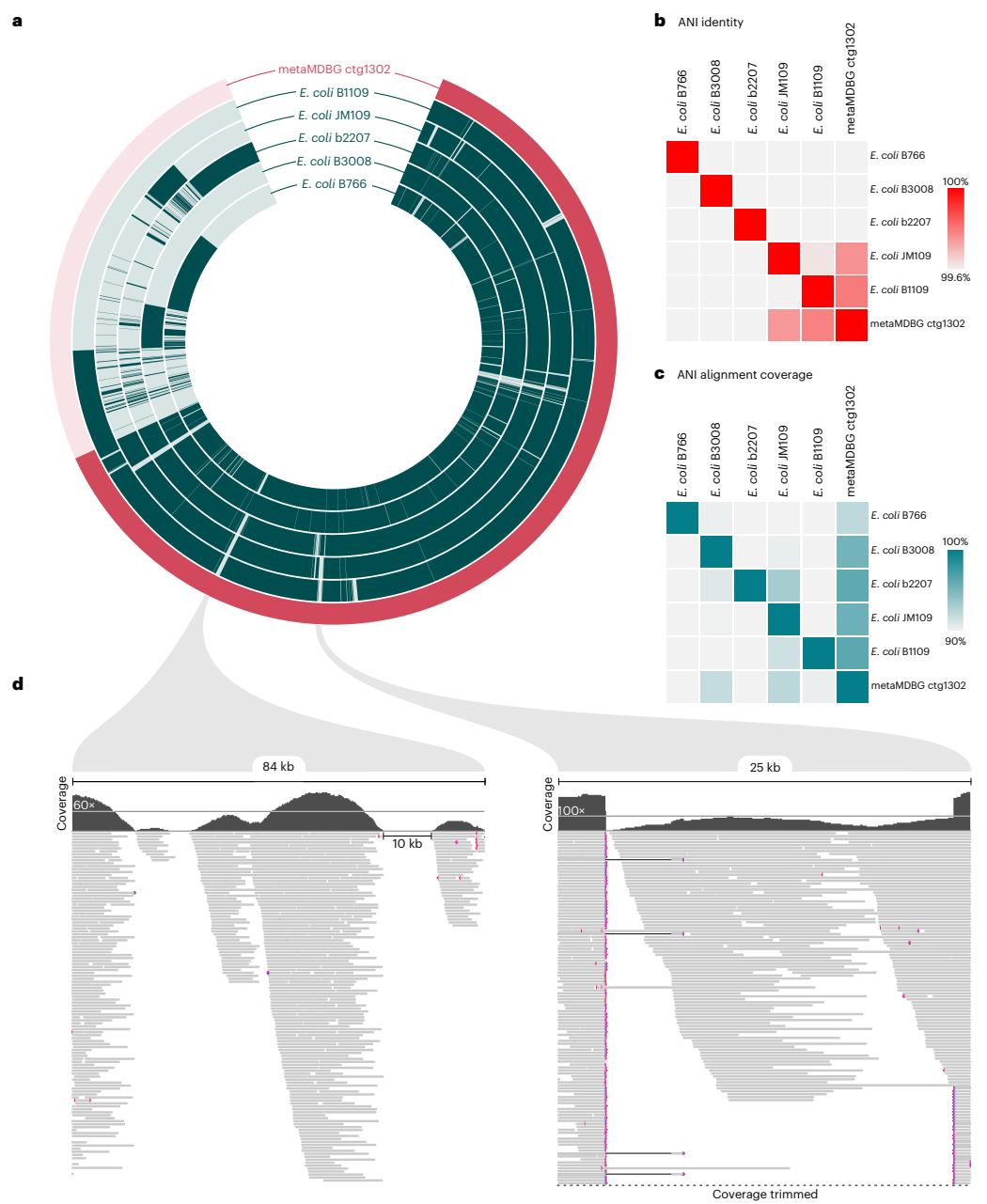
本研究建立的錯誤診斷框架已推動算法改進,metaMDBG v1.2和新型工具myloasm通過整合讀段剪切分析顯著降低錯誤率。研究人員強調,組裝算法應加強基于輸入讀段的后期糾錯,并提供可調節的啟發式參數以滿足不同精度需求。該工作為長讀長宏基因組學時代的基因組重建可靠性樹立新標準,對生物技術、生物醫學及公共基因組數據庫質量保障具有深遠意義。
 生物通微信公眾號
生物通微信公眾號
知名企業招聘